2.2.8 研究 | 2021-多组学分析肺结核队列的记忆T细胞状态
刘小泽写于2021.5.25
速览
Platforms GPL11154 Illumina HiSeq 2000 (Homo sapiens) Samples GSM4810298 Memory_T_cell_CITE-seq GSE158769_exprs_norm.tsv.gz 3.0 Gb (ftp)(http) TSV GSE158769_exprs_raw.tsv.gz 747.0 Mb (ftp)(http) TSV GSE158769_meta_data.txt.gz 19.8 Mb (ftp)(http) TXT
摘要
方法
先用CITE-seq测了>500,000个T细胞
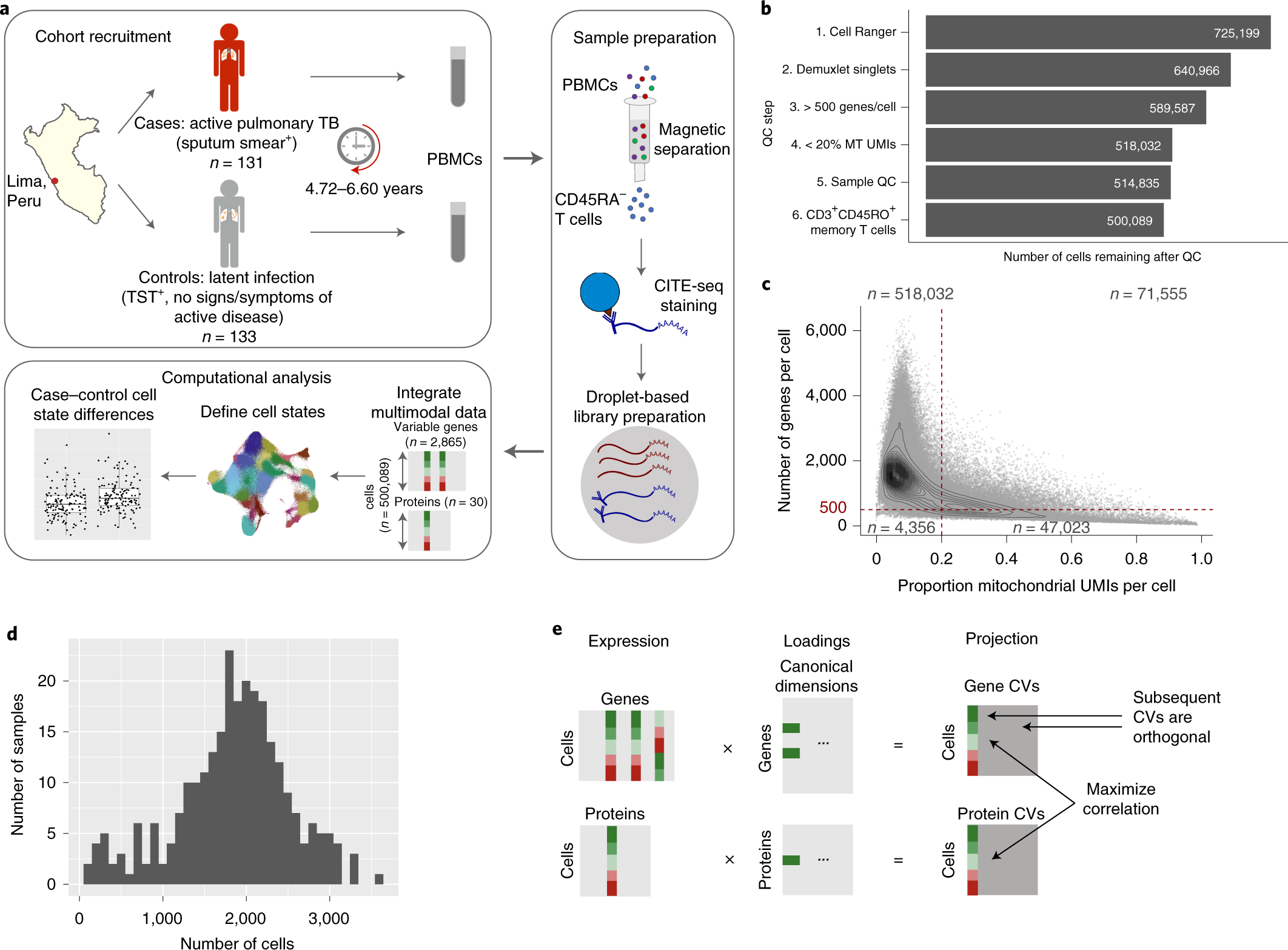
得到记忆T细胞的31种不同状态

记忆T细胞的状态因人口特征和环境而异
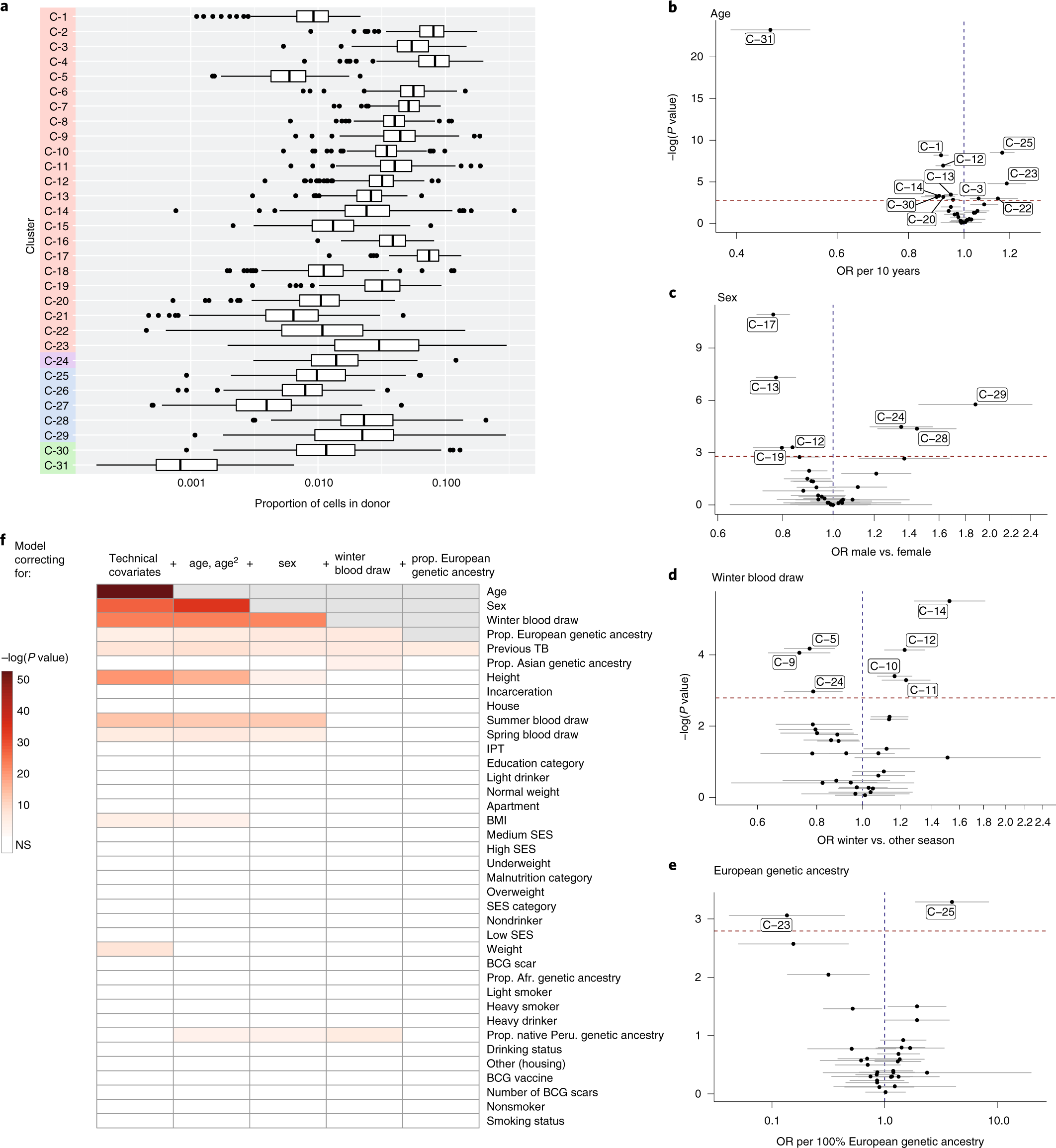
之后发现一个感兴趣的C-12状态



最后更新于

